Enrichissement des cibles
Les génomes des insectes et des agents pathogènes peuvent être complexes à décoder. Afin de faciliter les analyses et l’interprétation des données génomiques des ennemis des forêts ciblés par le projet bioSAFE, nous développons une approche d'enrichissement qui vise et séquence spécifiquement les régions du génome qui sont utiles pour répondre aux questions pertinentes à l'identification de leur espèce ou sous-espèce, leur origine et leurs traits. La plate-forme bioSAFE utilise une approche basée sur une amplification hautement multiplexée qui convient à la manipulation d'échantillons environnementaux d'origine inconnue et d’ états de conservation divers. Le séquençage de nouvelle génération est utilisé pour identifier des changements spécifiques dans l'ADN en séquençant plusieurs gènes ciblés dans plusieurs échantillons en une seule réaction. Cette approche est puissante, précise et se prête au traitement à haut débit. Elle est largement utilisée pour la détection du cancer dans le domaine médical. Notre application de cette approche pour la biosurveillance génomique des ennemis des forêts fournira un ensemble puissant d'outils pour la détection et la surveillance des espèces exotiques envahissantes.
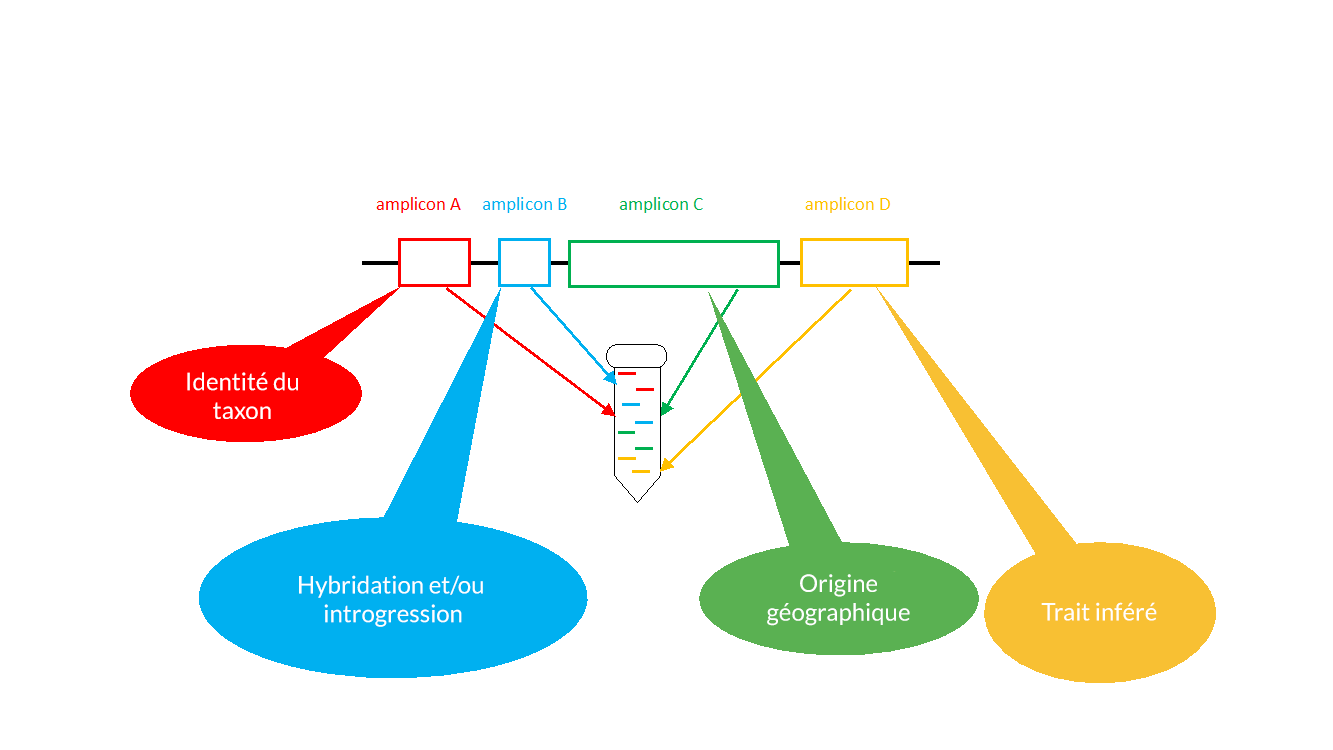
Figure 1. Exemple d'un panel d'enrichissement Ampliseq qui vise à séquencer plusieurs régions du génome pour répondre à différentes questions.
Comment la technologie sera-elle utilisée dans ce projet ?
Notre objectif est de concevoir des panels AmpliSeq (ThermoFisher) avec plusieurs couches d'amplicons (courts fragments d'ADN) qui ciblent différentes régions génomiques d'intérêt. Par exemple, un panel pourra contenir des régions du génome qui sont conservées dans plusieurs taxons et informatives sur le plan taxonomique en plus de contenir d'autres régions que nous avons identifiées comme étant fréquemment impliquées dans l'hybridation adaptative et l'introgression (figure 1). Une autre couche pourra contenir des amplicons qui sont hautement polymorphes au sein des espèces et qui nous renseignent sur l'origine géographique et les voies de propagation. Une dernière couche pourra contenir des régions du génome qui sont associées à des caractères considérées comme importantes pour le caractère invasif telles que la capacité de vol, la tolérance au froid, la pathogénicité et la gamme d'hôtes (figure 1).
La boîte à outils bioSAFE
Un panel sera conçu pour chacun des ravageurs ou agents pathogènes. Actuellement, nous avons développé le panel Phytoseq qui cible plus de 30 régions du génome de Phytophthora et permet une amplification de 6000 pb (nucléotides) dans des groupes de 96 individus. Cet outil puissant permet à l'utilisateur d'obtenir des données sur la séquence du génome directement à partir d'échantillons environnementaux et/ou de cultures pures et ainsi d’associer des échantillons inconnus à un clade de Phytophthora, à une espèce et même à une lignée (figure 2).

Figure 2. Amplification et séquençage des régions ciblées du génome directement à partir des tissus végétaux infectés et attribution précise aux lignées de P. ramorum.

